Práctico 2 Supuestos de ANOVA y mínimos cuadrados
2.1 Paquetes necesarios para este práctico
Para este capitulo necesitas tener instalado el paquete tidyverse, también ayuda tener el paquete broom. Esta clase del curso puede también ser seguida en este link. El video de la clase se encuentra disponible en este link.
2.2 Objetivos de este práctico
- Entender los supuestos de un ANOVA de una vía (independencia, aleatoriedad, homocedasticidad y normalidad)
- Entender el concepto de mínimos cuadrados
- Saber cuando realizar un ANOVA e interpretar sus resultados
2.3 Actividad 1 Sueño en mamíferos
En esta actividad intentaremos ver si hay diferencias en horas de sueño en mamíferos por Orden o dieta. Los datos fueron extraídos del trabajo de Savage and West (2007) y están incorporados en la base de datos de ggplot2 con el nombre de msleep, pero estarán en webcursos en formato csv de todas formas. Para la guía los ejemplos se generarán en base a la base de datos InsectSprays que está en R y que fue extraída de Beall (1942), en la cual se testean la efectividad de insecticidas en Spray en la abundancia de insectos en plantaciones. Y en la base de datos iris que ya fue entregada, en la que se miden distintas características florales de especies del genero Iris (Anderson 1935).
2.3.1 Homogeneidad de varianza
2.3.1.1 Inspección visual
Lo primero que intentaremos explorar de forma visual y a partir de tests si es que hay homogeneidad de varianza, para esto usaremos boxplots, y jitter plots (Figura 2.1), lo cual ya hemos hecho anteriormente:
ggplot(InsectSprays, aes(x = spray, y = count)) + geom_boxplot() + geom_jitter(aes(color = spray)) 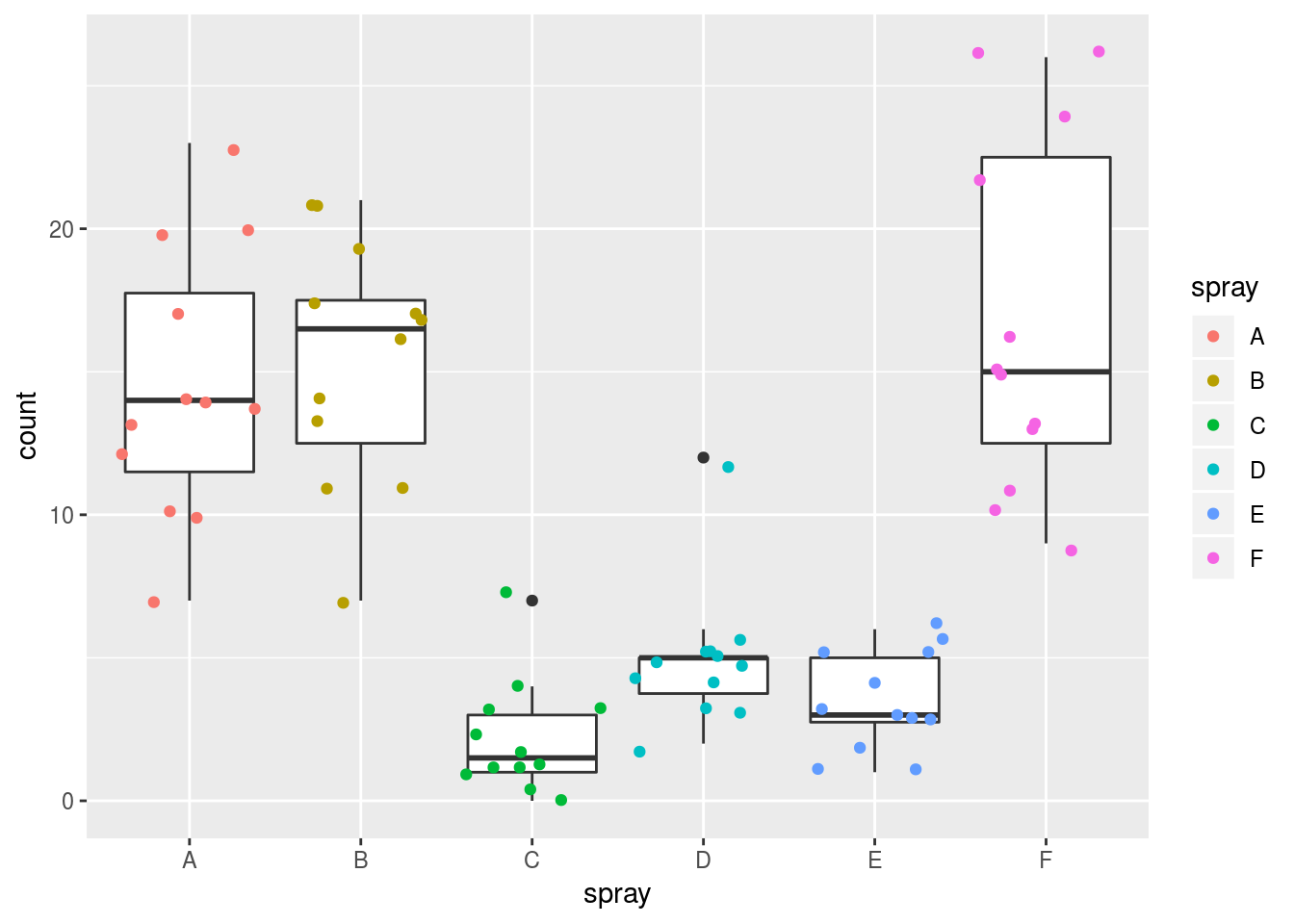
Figura 2.1: Cuenta de insectos según tipo de insecticida
Para explorar visualmente si existe homogeneidad de varianza, se compraran las cajas y bigotes de los boxplots y se espera que tengan (Mas o menos distintos tamaños).
2.3.1.2 Test de Bartlett
Para realizar un test de homogeneidad de varianza se realiza el test de bartlett (Bartlett 1937), en este se usa nuestra conocida formula y ~ x, esto es, y explicado por x junto a la función bartlett.test. Para nuestro caso usaríamos:
##
## Bartlett test of homogeneity of variances
##
## data: count by spray
## Bartlett's K-squared = 25.96, df = 5, p-value = 9.085e-05Como en este caso, no el valor de p es menor a 0.05, decimos que no hay homogeneidad de varianza, por lo que no podemos hacer el test.
2.3.2 Normalidad de los residuales
En el caso de la base de datos iris, demostraremos inmediatamente que si hay homogeneidad de varianza en el ancho del sépalo (Figura 2.2):
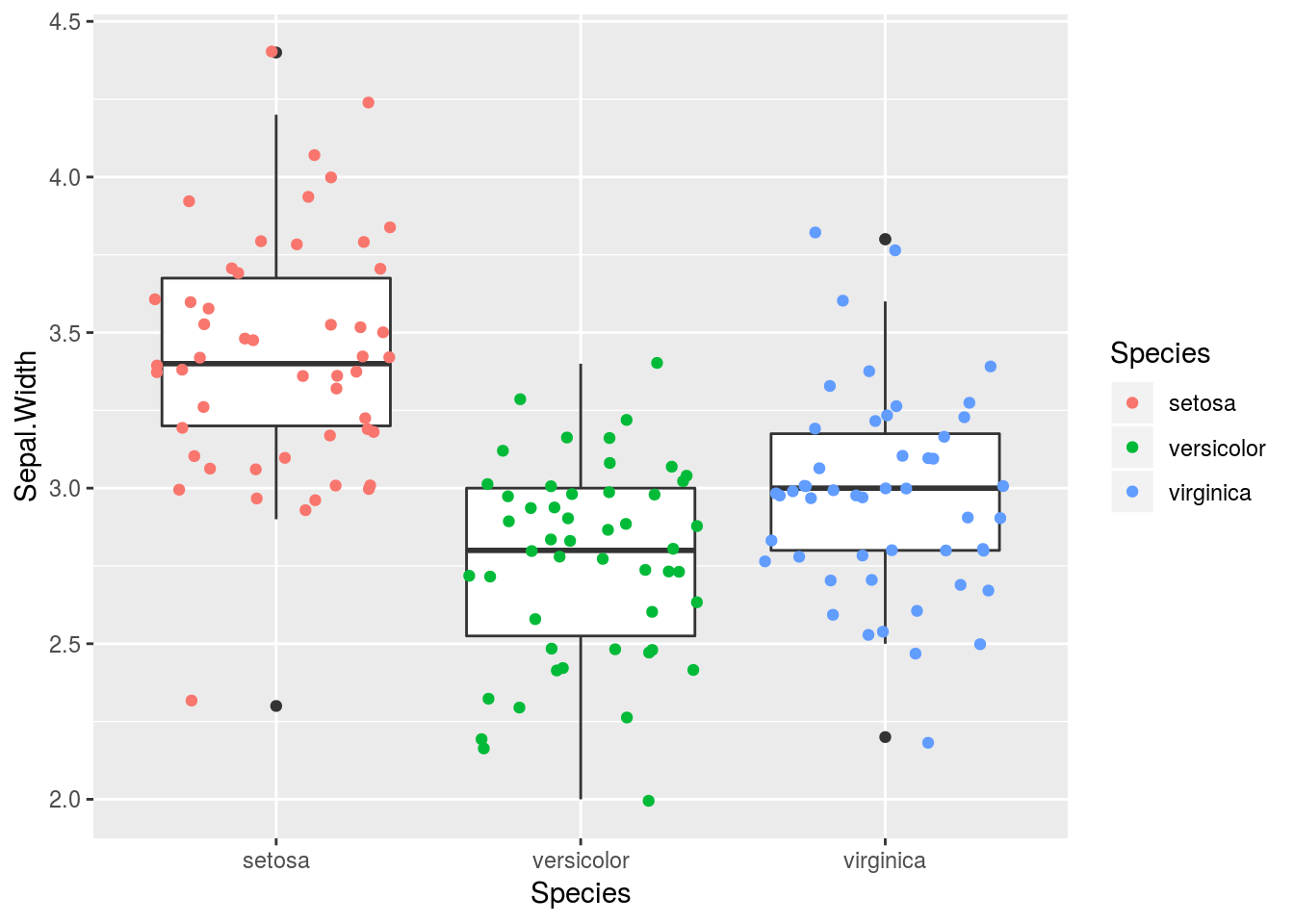
Figura 2.2: Ancho de sépalo según especie del género Iris
##
## Bartlett test of homogeneity of variances
##
## data: Sepal.Width by Species
## Bartlett's K-squared = 2.0911, df = 2, p-value = 0.3515Debido a ello, podemos testar si los residuales tienen una distribución normalidad de los residuales, para esto lo primero que debemos hacer es un ANOVA, como fue explicado en el práctico anterior y guardar este objeto con un nombre:
2.3.2.1 Extracción de los residuales del modelo
Para extraer los residuales, podemos hacerlo de dos formas, si solo queremos un vector de sus valores, podemos extraerlo desde el modelo mismo utilizando $residuals. Si queremos guardarlo en un dataframe mas completo podemos utilizar la función augment del paquete broom.
La segunda opción nos entregará más información que podremos utilizar más tarde, pero ambas sirven para testear normalidad, la siguiente tabla muestra las primeras 6 observaciones generadas por la función augment, donde resid, son los residuales (Ver tabla 2.1.
| Sepal.Width | Species | .fitted | .se.fit | .resid | .hat | .sigma | .cooksd | .std.resid |
|---|---|---|---|---|---|---|---|---|
| 3.5 | setosa | 3.428 | 0.048 | 0.072 | 0.02 | 0.341 | 0.000 | 0.214 |
| 3.0 | setosa | 3.428 | 0.048 | -0.428 | 0.02 | 0.339 | 0.011 | -1.273 |
| 3.2 | setosa | 3.428 | 0.048 | -0.228 | 0.02 | 0.340 | 0.003 | -0.678 |
| 3.1 | setosa | 3.428 | 0.048 | -0.328 | 0.02 | 0.340 | 0.006 | -0.975 |
| 3.6 | setosa | 3.428 | 0.048 | 0.172 | 0.02 | 0.341 | 0.002 | 0.511 |
| 3.9 | setosa | 3.428 | 0.048 | 0.472 | 0.02 | 0.339 | 0.013 | 1.404 |
2.3.2.2 Inspección visual de los residuales
Existen dos formas de visualizar los residuales para determinar si la distribución de estos es o no es normal, histogramas y el qqplot.
2.3.2.2.1 Histograma
Los histogramas nos darán una representación visual para tratar de entender si la distribución es normal, para esto, solo necesitamos usar el comando hist, seguido del vector de los residuales, este es el comando para hacer el histograma (Figura 2.3) con cualquiera de las dos bases de datos, el resultado debiera ser el mismo:
hist(Residuales)
hist(Resultados$.resid)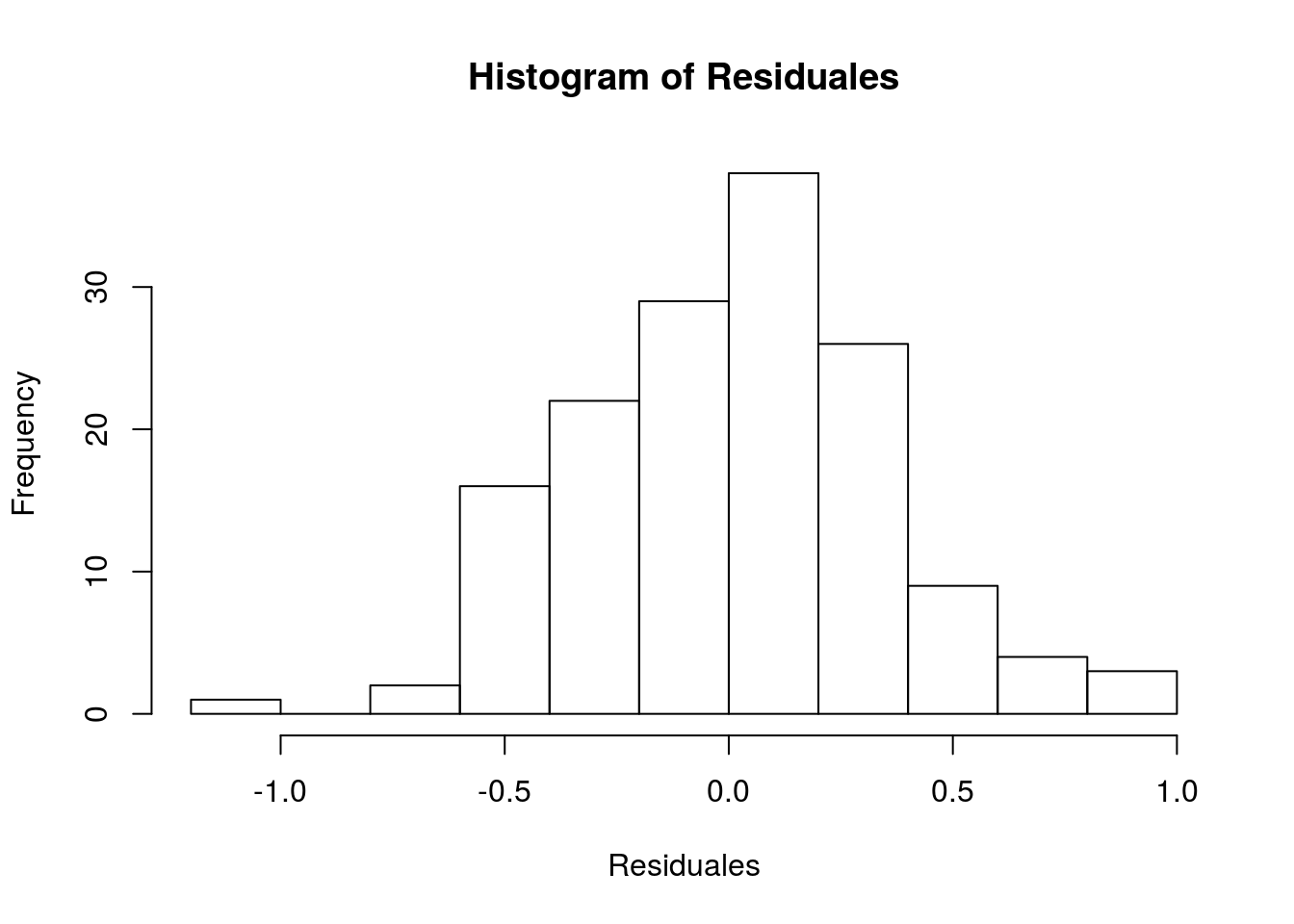
Figura 2.3: Histograma de los resiudales del modelo ANOVA
2.3.2.2.2 QQplot
El qq plot es otra forma visual de establecer si los residuales son o no son normales, para esto, lo esperado es que la gráfica resultante sea una diagonal lo mas recta posible, para esto usaremos la función qqnorm, con nuestros residuales, de nuevo, podemos usar cualquiera de las dos versiones de nuestros datos:
qqnorm(Residuales)
qqnorm(Resultados$.resid)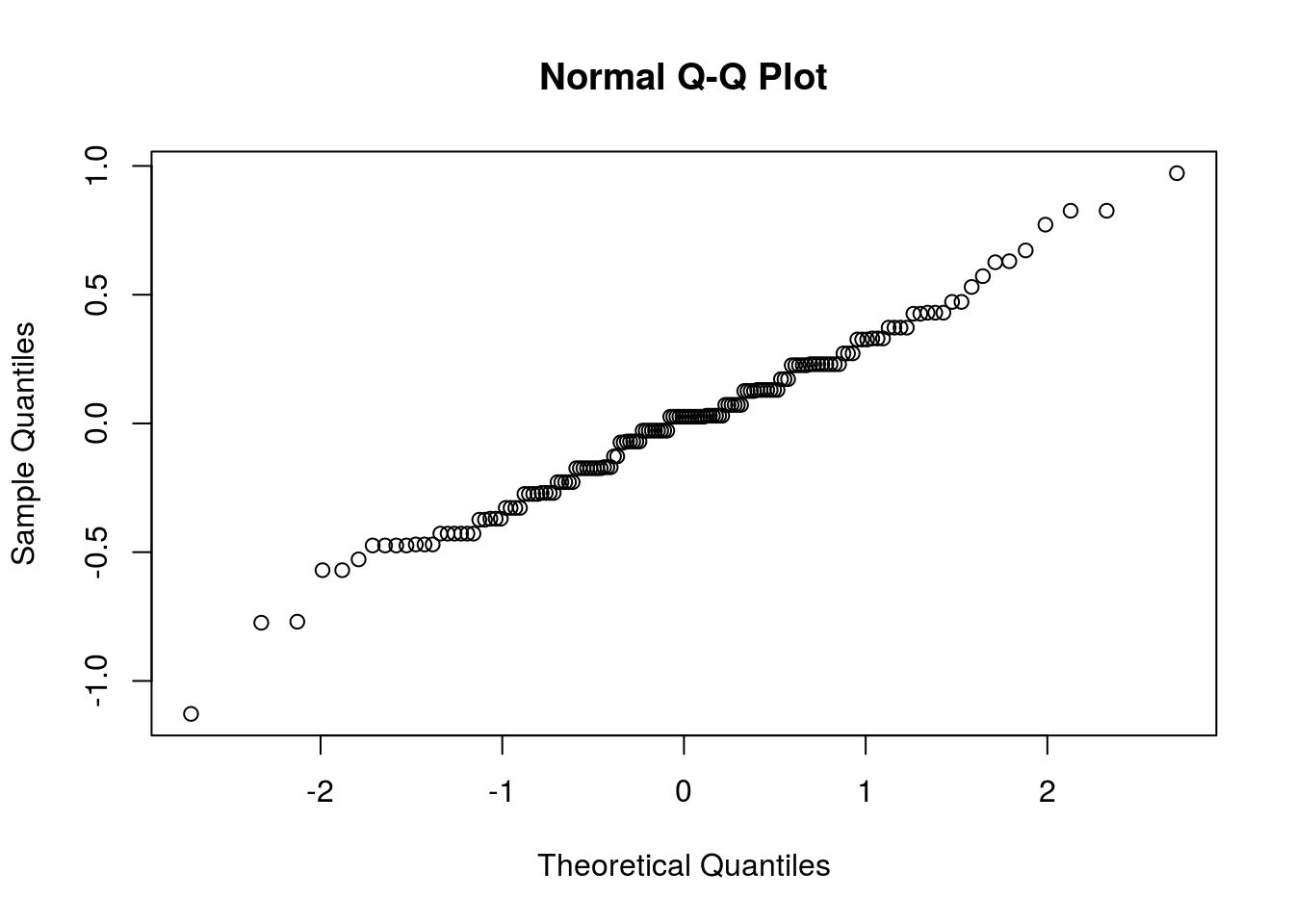
Figura 2.4: qqplot de los resiudales del modelo ANOVA
2.3.2.3 Test de Shapiro para determinar normalidad
La forma más sencilla de determinar normalidad es usando el test de Shapiro-Wilk de normalidad (Royston 1995). Al igual que el test de Bartlett, si el valor de p es menor a 0.05, determinamos que la distribución de los datos no son normales, la función en R para este test es shapiro.test, y al igual que en los casos anteriores de hist y qqpot, solo necesitamos de usar un vector de residuales para ver el resultado del test. En nuestro caso:
shapiro.test(Residuales)
shapiro.test(Resultados$.resid)##
## Shapiro-Wilk normality test
##
## data: Residuales
## W = 0.98948, p-value = 0.323Ya que el valor de p es menor a 0.05, podemos decir que la distribución de nuestros residuales es normal, y por lo tanto el test cumple con los supuestos, y esto hace que sea valido el ANOVA, por lo que podemos ver nuestros resultados. La homogeneidad de Varianza es mas importante que la normalidad de residuales para estos casos, para ejemplos de lo que se debe hacer si se viola la normalidad ver Lix, Keselman, and Keselman (1996)
2.4 Actividad 2 Suma de cuadrados
Tanto los ANOVAS como las regresiones lineales se basan en minimizar la suma de cuadrados, es la suma de los cuadrados de los errores o residuales.
2.4.1 ¿Que es el error? ¿Por qué al cuadrado??
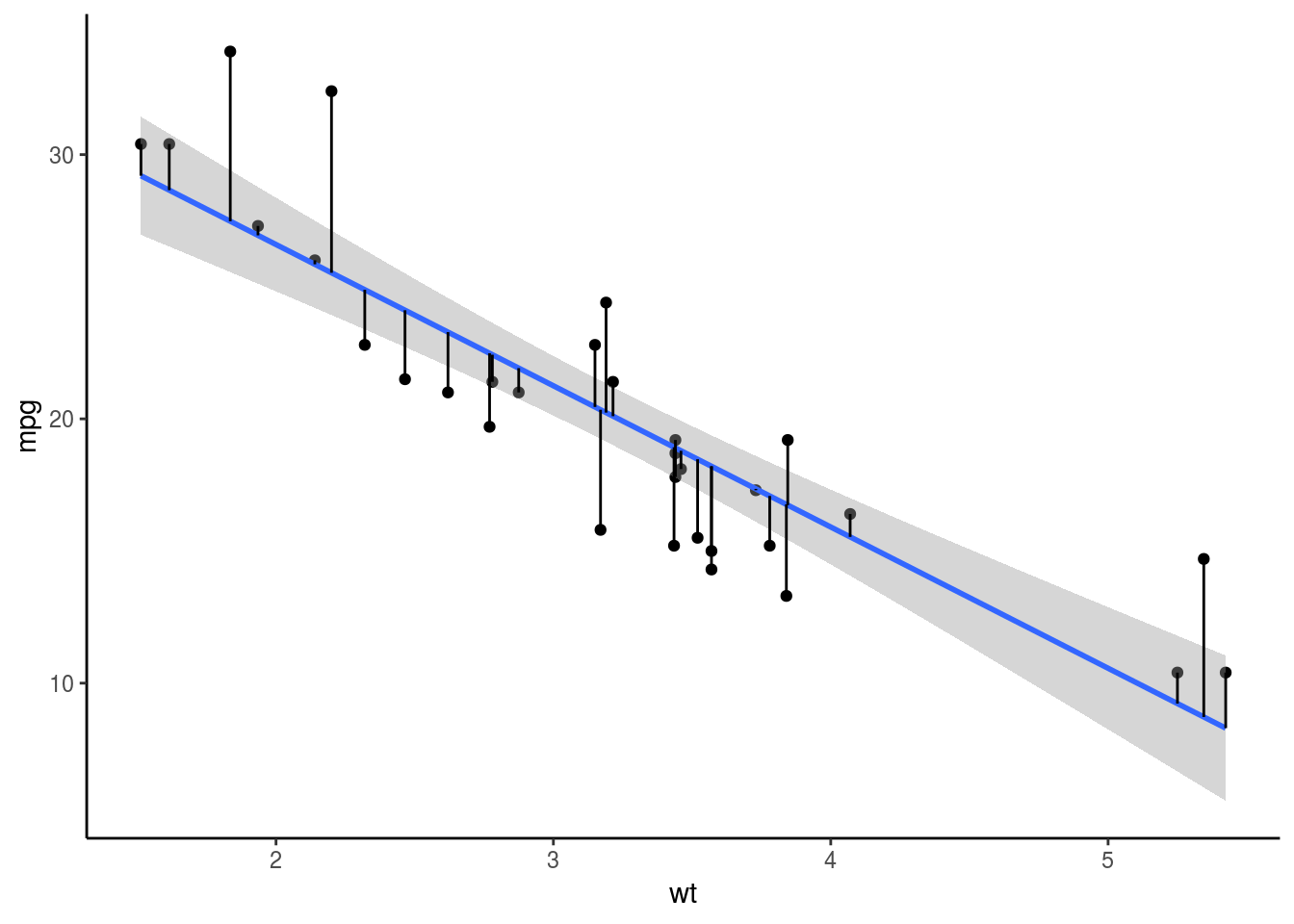
Figura 2.5: Errores de una regresión lineal ejemplificados con la linea entre el valor predicho y el observado
En la figura y en la formula vemos ejemplificado que es el error, también conocido como residual, este es simplemente el valor observado
\[Observado - Predicho\]
El objetivo de todo modelo es el de minimizar estos errores, al ajustar el mejor modelo posible.
Los errores siempre se calculan al cuadrado, discutiremos por que en clase
\[\sum_{i=1}^{n} (Observado - Predicho)^2\]
2.5 Referencias
Referencias
Savage, Van M, and Geoffrey B West. 2007. “A Quantitative, Theoretical Framework for Understanding Mammalian Sleep.” Proceedings of the National Academy of Sciences 104 (3). National Acad Sciences: 1051–6.
Beall, Geoffrey. 1942. “The Transformation of Data from Entomological Field Experiments so That the Analysis of Variance Becomes Applicable.” Biometrika 32 (3/4). JSTOR: 243–62.
Anderson, Edgar. 1935. “The Irises of the Gaspe Peninsula.” Bulletin of the American Iris Society 59: 2–5.
Bartlett, Maurice S. 1937. “Properties of Sufficiency and Statistical Tests.” Proc. R. Soc. Lond. A 160 (901). The Royal Society: 268–82.
Royston, Patrick. 1995. “Remark as R94: A Remark on Algorithm as 181: The W-Test for Normality.” Journal of the Royal Statistical Society. Series C (Applied Statistics) 44 (4). JSTOR: 547–51.
Lix, Lisa M, Joanne C Keselman, and HJ Keselman. 1996. “Consequences of Assumption Violations Revisited: A Quantitative Review of Alternatives to the One-Way Analysis of Variance F Test.” Review of Educational Research 66 (4). Sage Publications Sage CA: Thousand Oaks, CA: 579–619.